2024年5月(yuè)14日,上海交通(tōng)大(dà)學生命科學技術學院餘祥長(cháng)聘教軌副教授課題組和(hé)辰山植物(wù)園楊俊/王紅霞團隊合作在國際知名期刊《Nature Communications》發表題爲“Transfer learning enables identification of multiple types of RNA modifications using nanopore direct RNA sequencing”的(de)研究論文。作者結合納米孔RNA直接測序技術和(hé)深度學習(xí)方法開發了(le)可(kě)同時(shí)檢測多(duō)種類型RNA修飾的(de)遷移學習(xí)模型TandemMod(圖1)。上海交通(tōng)大(dà)學生命科學技術學院博士後吳優爲論文第一作者,上海交通(tōng)大(dà)學生命科學技術學院餘祥長(cháng)聘教軌副教授、辰山植物(wù)園王紅霞副研究員(yuán)和(hé)楊俊研究員(yuán)爲該論文通(tōng)訊作者。
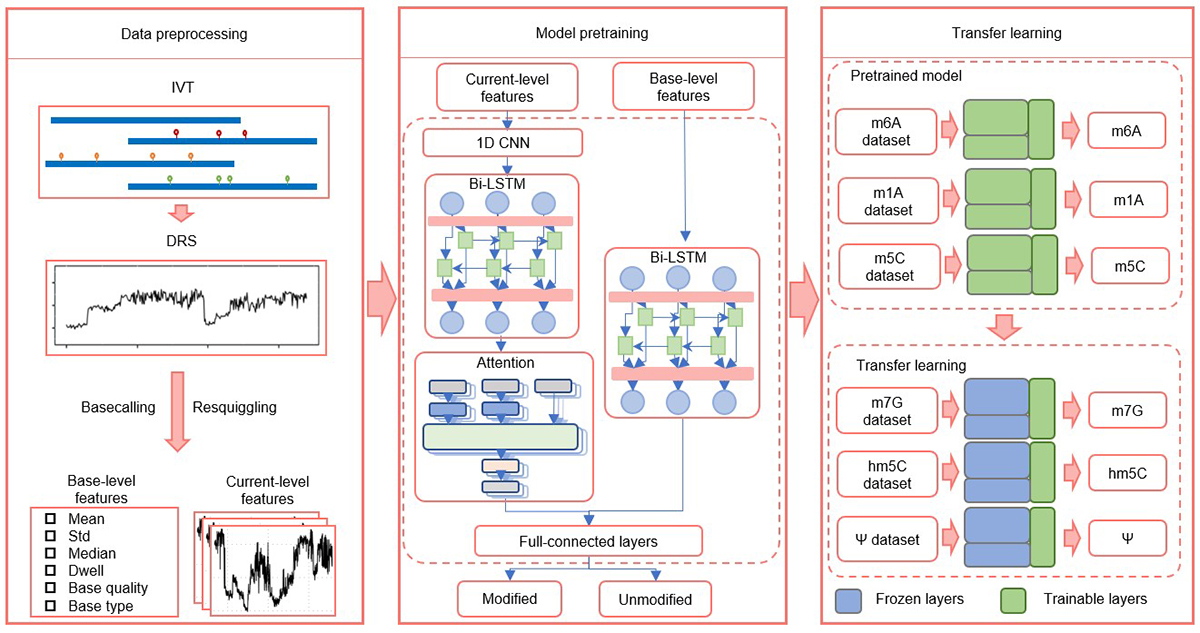
圖1. 基于納米孔RNA直接測序數據搭建TandemMod深度學習(xí)框架的(de)示意圖。
自然界RNA中存在160種以上的(de)修飾類型。近年的(de)研究揭示mRNA上的(de)m6A、m5C、m1A和(hé)Ψ等修飾具有重要的(de)生物(wù)學功能。結合抗體捕獲、化(huà)學突變和(hé)限制性酶切等方法的(de)高(gāo)通(tōng)量測序技術被廣泛用(yòng)于對(duì)mRNA某一類型修飾位點的(de)鑒定。然而,如何在同一條mRNA轉錄本中同時(shí)鑒定多(duō)種不同類型的(de)修飾位點還(hái)存在巨大(dà)挑戰。相較于二代測序技術,納米孔RNA直接測序 (Direct RNA Sequencing, DRS)能夠對(duì)帶有polyA尾的(de)全長(cháng)mRNA進行直接測序,mRNA穿過納米孔時(shí)産生的(de)電信号差異可(kě)用(yòng)于推斷單條mRNA上每個(gè)堿基的(de)類别及其修飾信息(圖2)。因此,結合機器學習(xí)算(suàn)法,理(lǐ)論上DRS可(kě)實現在單條mRNA水(shuǐ)平、以單堿基分(fēn)辨率檢測多(duō)種不同類型的(de)堿基修飾。但是,目前納米孔的(de)電信号特征與天然mRNA上的(de)哪些修飾堿基相對(duì)應還(hái)缺乏準确的(de)标簽,這(zhè)制約了(le)RNA修飾檢測模型的(de)訓練和(hé)建立。
圖2. RNA通(tōng)過納米孔時(shí)産生電信号信息的(de)示意圖。
該研究中作者創新性地利用(yòng)帶有T7啓動子的(de)水(shuǐ)稻cDNA文庫體外轉錄出了(le)具備不同修飾标簽的(de)數千種mRNA轉錄本,加polyA尾後通(tōng)過DRS創建了(le)分(fēn)别帶有未修飾堿基、m6A修飾、m5C修飾和(hé)m1A修飾等精準标簽的(de)體外表觀轉錄組 (IVET)數據集(圖3)。在此基礎上,作者以每5個(gè)堿基分(fēn)配到的(de)電信号及其統計特征作爲輸入,訓練并搭建了(le)一個(gè)具備1維卷積神經網絡 (1D CNN)、雙向長(cháng)短時(shí)記憶(Bi-LSTM)和(hé)注意力機制(Attention)的(de)TandemMod深度學習(xí)框架(圖1)。
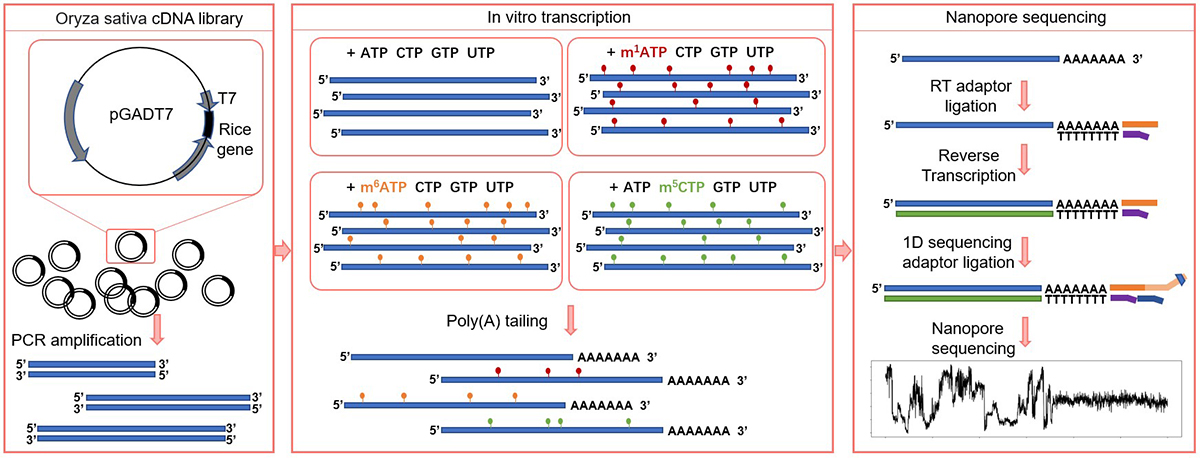
圖3. 利用(yòng)水(shuǐ)稻cDNA文庫體外轉錄帶有m6A、m1A和(hé)m5C标簽的(de)轉錄本、并通(tōng)過DRS創建體外表觀轉錄組的(de)示意圖。
作者首先将TandemMod模型與目前基于DRS預測m6A和(hé)m5C的(de)主流算(suàn)法進行比較,驗證了(le)TandemMod模型在體外轉錄本及細胞體内轉錄本DRS數據中預測RNA修飾位點的(de)優越性能(圖4)。在正常人(rén)類細胞中檢測到排名前五的(de)基序中有4個(gè)是已知的(de)DRACH,在m6A甲基化(huà)酶敲除的(de)細胞中m6A修飾率顯著下(xià)降。
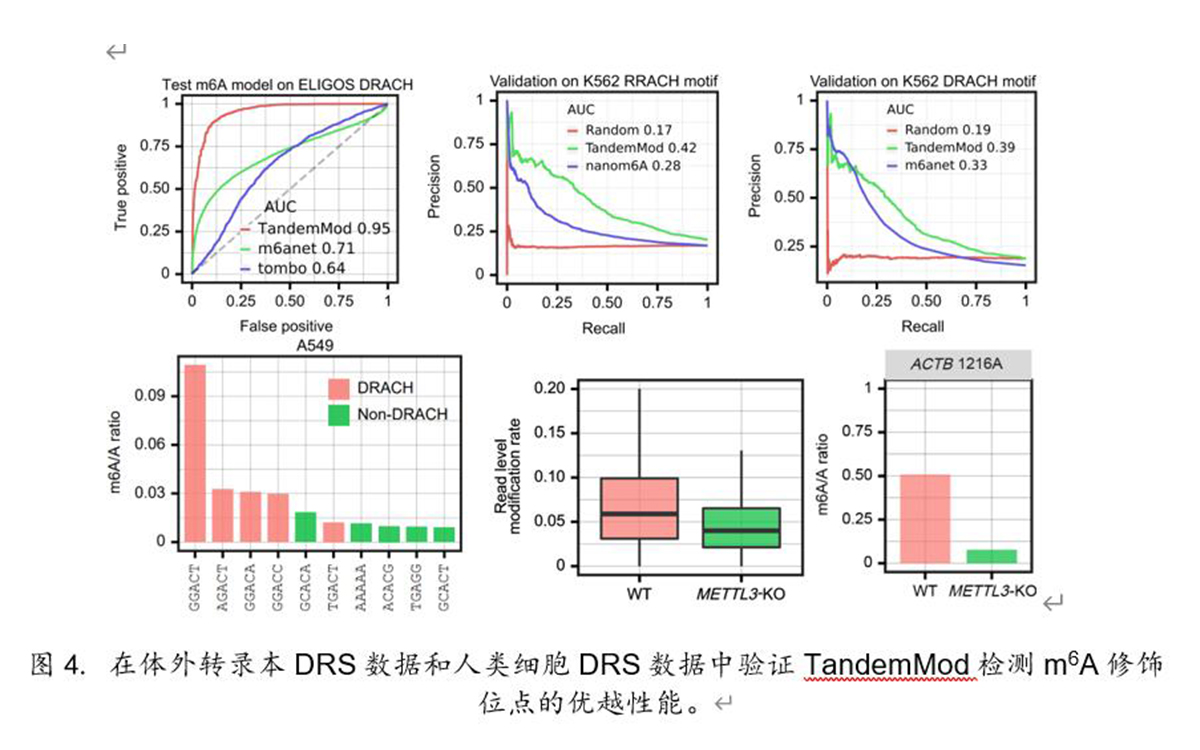
圖4. 在體外轉錄本DRS數據和(hé)人(rén)類細胞DRS數據中驗證TandemMod檢測m6A修飾位點的(de)優越性能。
進一步,作者通(tōng)過遷移學習(xí)将TandemMod用(yòng)于檢測mRNA中的(de)m7G、 hm5C、 Ψ 和(hé)Inosine等多(duō)種類型的(de)RNA修飾位點,揭示了(le)遷移學習(xí)可(kě)在保證同等性能的(de)條件下(xià)顯著減少訓練集數據量和(hé)模型訓練時(shí)間等計算(suàn)成本(圖5)。最後,作者通(tōng)過TandemMod繪制了(le)水(shuǐ)稻幼苗在高(gāo)鹽脅迫下(xià)m6A、m5C和(hé)Ψ的(de)表觀修飾圖譜,并揭示了(le)mRNA中m6A和(hé)m5C的(de)共修飾及其在高(gāo)鹽環境下(xià)的(de)修飾率變化(huà)(圖6)。
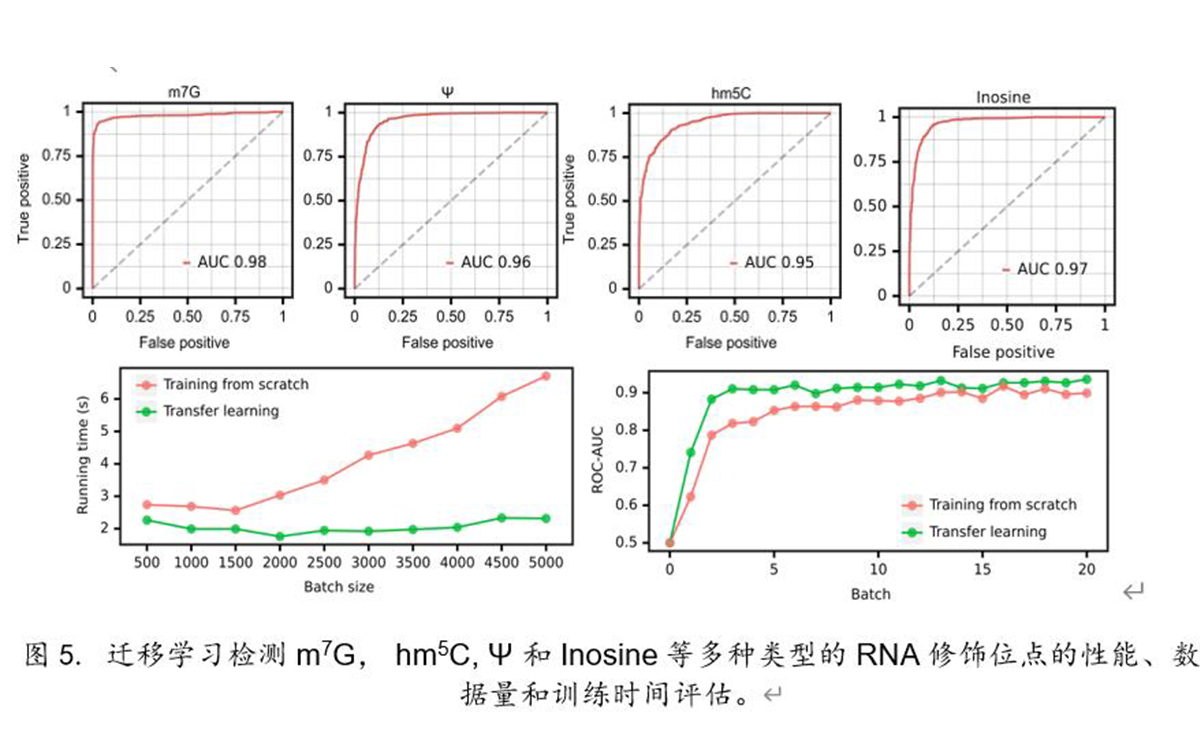
圖5. 遷移學習(xí)檢測m7G, hm5C, Ψ 和(hé)Inosine等多(duō)種類型的(de)RNA修飾位點的(de)性能、數據量和(hé)訓練時(shí)間評估。
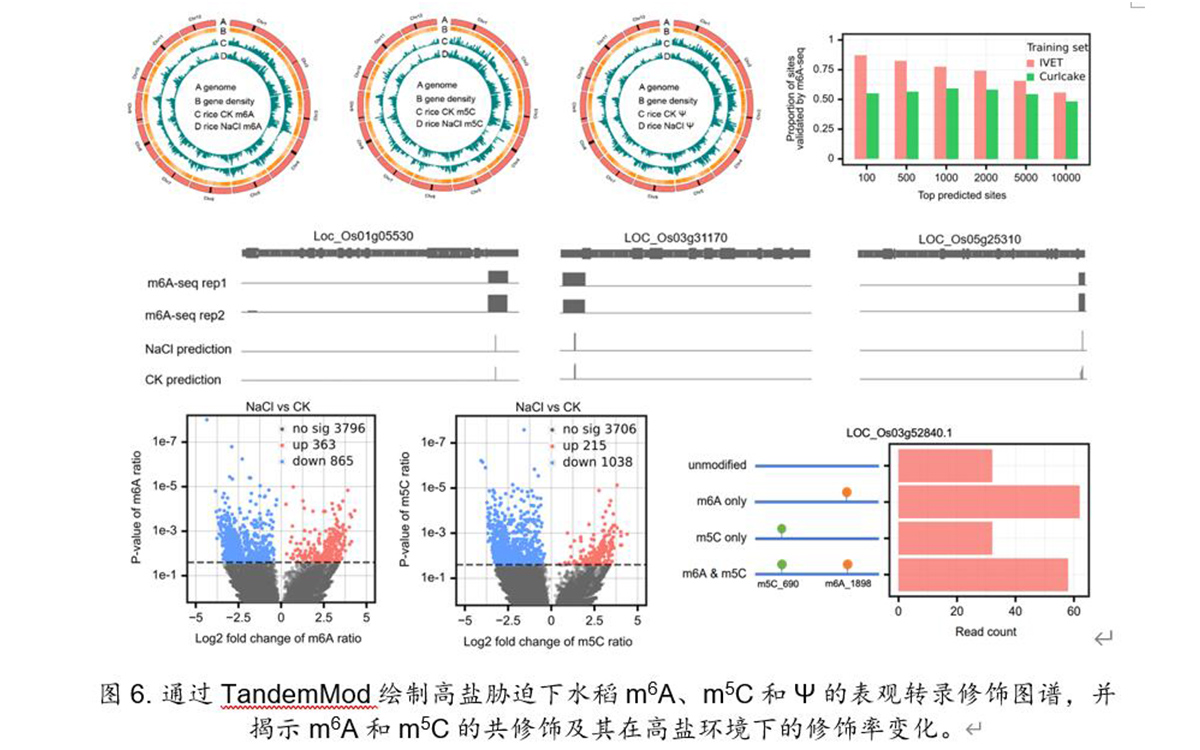
圖6. 通(tōng)過TandemMod繪制高(gāo)鹽脅迫下(xià)水(shuǐ)稻m6A、m5C和(hé)Ψ的(de)表觀轉錄修飾圖譜,并揭示m6A和(hé)m5C的(de)共修飾及其在高(gāo)鹽環境下(xià)的(de)修飾率變化(huà)。
綜上,作者創建的(de)體外表觀轉錄組 (IVET) 将爲基于納米孔RNA單分(fēn)子測序鑒定RNA修飾的(de)機器學習(xí)方法提供基準訓練數據集,開發的(de)可(kě)遷移深度學習(xí)模型TandemMod将爲動植物(wù)和(hé)微生物(wù)體内多(duō)種類型的(de)RNA修飾位點鑒定及表觀轉錄組研究提供重要的(de)技術支撐。同時(shí)TandemMod還(hái)可(kě)用(yòng)于檢測RNA疫苗等人(rén)工修飾的(de)RNA。
該研究得(de)到了(le)國家自然科學基金、上海市浦江人(rén)才和(hé)上海市科技創新行動計劃的(de)支持。感謝上海辰山植物園研究平台在納米孔測序的(de)幫助以及上海交通(tōng)大(dà)學超算(suàn)平台提供的(de)計算(suàn)資源。
論文鏈接:https://www.nature.com/articles/s41467-024-48437-4
文:王紅霞